Publikujemy w eLife!
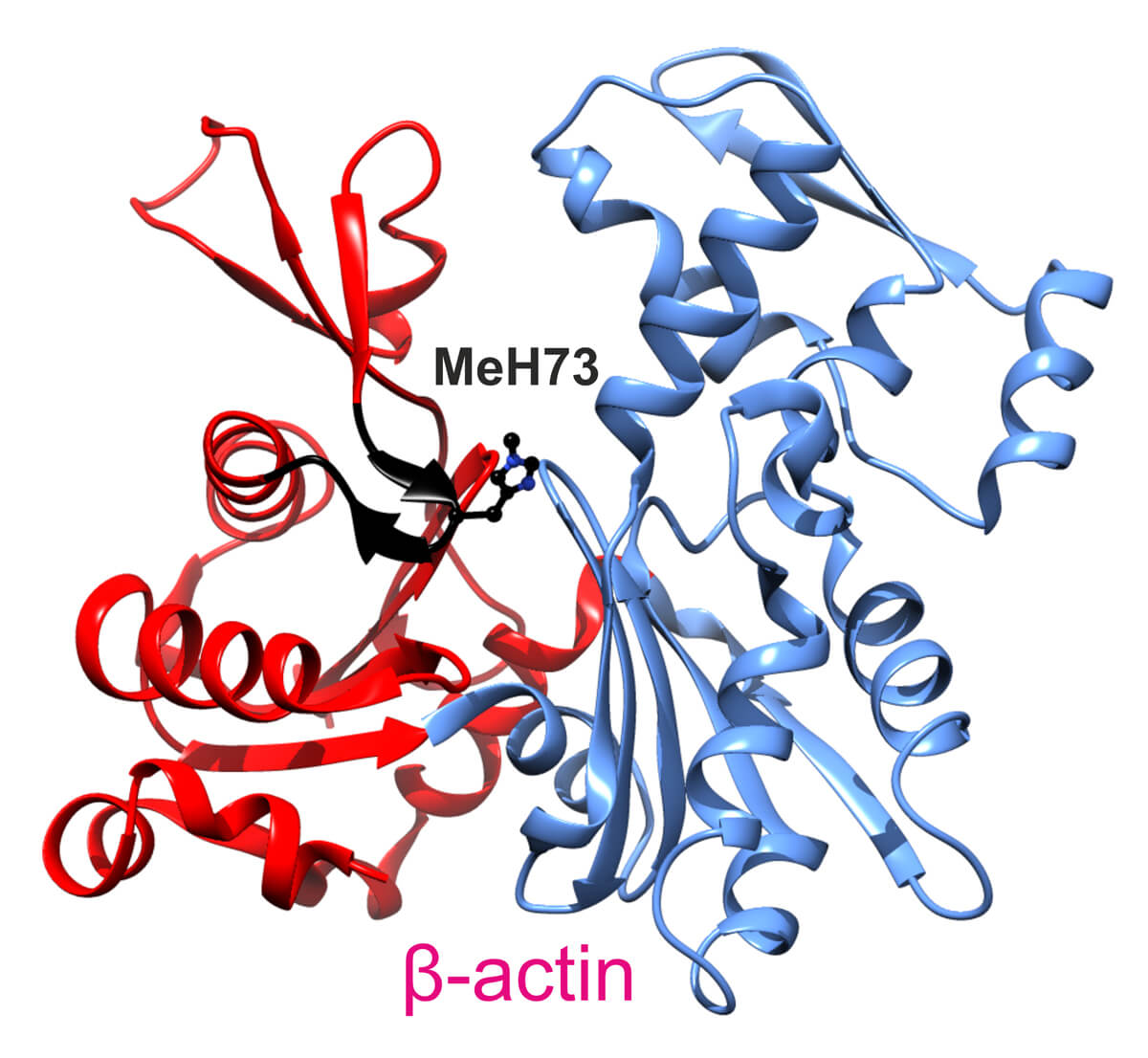
Zespół naukowców z Wydziału Biologii w składzie: Sebastian Kwiatkowski, Agnieszka Seliga, Takao Ishikawa, Iwona Grabowska oraz Adam Jagielski, kierowany przez Jakuba Drożaka, we współpracy z naukowcami z Belgii oraz Niemiec, zidentyfikował gen kodujący jeden z najbardziej tajemniczych enzymów sierocych występujących u ssaków. Zarówno tożsamość molekularna (gen), jak również funkcja biologiczna N-metylotransferazy histydynowej aktyny (EC 2.1.1.85) pozostawały nieznane od 1987 roku, kiedy to pierwszy raz opisano jej aktywność w organizmie królika. Po 21 latach od opublikowania pierwszej pracy opisującej badany enzym, publikacja Zespołu z UW dostarcza odpowiedź, co do jego natury molekularnej.
Autorzy pracy SETD3 protein is the actin-specific histidine N-methyltransferase(https://doi.org/10.7554/eLife.37921) zdołali oczyścić enzym z tkanek szczura oraz zidentyfikować jego gen wykorzystując sekwencjonowanie białek metodą spektrometrii mas. Otrzymane wyniki pozwoliły na identyfikację ludzkiego enzymu oraz jego charakterystykę biochemiczną oraz fizjologiczną.
Wyniki opublikowane na łamach czasopisma eLife rzucają nowe światło na specyficzność substratową enzymów z rodziny SET, których większość (w tym również SETD3) jest obecnie klasyfikowana jako N-metylotransferazy lizynowe, modyfikujące potranslacyjnie łańcuchy boczne histonów. Zidentyfikowana N-metylotransferaza jest pierwszym ssaczym przedstawicielem nowej klasy enzymów – metylotransferaz histydynowych białek.
Zespół kierowany przez Jakuba Drożaka kontynuuje badania nad białkiem SETD3, a także rozwija kolejne wątki badawcze w poszukiwaniu tożsamości molekularnych kolejnych enzymów sierocych ssaków.
