BADANIA NAUKOWCÓW Z IGIB W NATURE I NATURE COMMUNICATIONS
Podstawowe procesy związane z ekspresją ludzkiego genomu mitochondrialnego zostały opisane już w latach 70-tych i 80-tych ubiegłego wieku, mimo to wiele aspektów funkcjonowania genomu mitochondrialnego człowieka nadal pozostaje tajemnicą. Badania przeprowadzone w grupie kierowanej przez prof. Piotra Stępnia z IGiB UW doprowadziły do odkrycia kluczowych enzymów odpowiedzialnych za obróbkę i degradację ludzkiego RNA mitochondrialnego. W lipcu tego roku ukazały się dwie publikacje (Nature Communications oraz Nature), które są kontynuację tych badań.
Pracownicy i doktoranci m. in. Wydziału Biologii UW na łamach czasopisma Nature Communications opublikowali pracę opisującą degradację niekodujących RNA w mitochondriach człowieka (https://www.nature.com/articles/s41467-018-05007-9). RNA powstały w wyniku transkrypcji niekodujących obszarów ludzkiego genomu mitochondrialnego zawiera dużo nukleotydów guaninowych, dzięki czemu może on tworzyć stabilne struktury tzw. G-kwadrupleksy. Badania przeprowadzone pod kierunkiem dr. Romana Szczęsnego oraz prof. Andrzeja Dziembowskiego pozwoliły wykazać, że mitochondrialny degradosom – kompleks złożony z helikazy SUV3 oraz egzorybonukleazy PNPazy – jest wspomagany przez białko GRSF1 w degradacji RNA zawierającego G-kwadrupleksy. Białko GRSF1 wiąże G-kwadrupleksy, co prowadzi do zniszczenia tej struktury ułatwiając degradację RNA przez degradosom. Analiza porównawcza białek posiadających domenę RRM, do których należy GRSF1, oraz sekwencji genomów mitochondrialnych pozwoliła zaproponować, że pojawienie się GRSF1 w określonej grupie kręgowców ma najprawdopodobniej charakter adaptacyjny i związane jest z występowaniem/wykształceniem się genomów mitochondrialnych, które kodują transkrypty G-bogate.
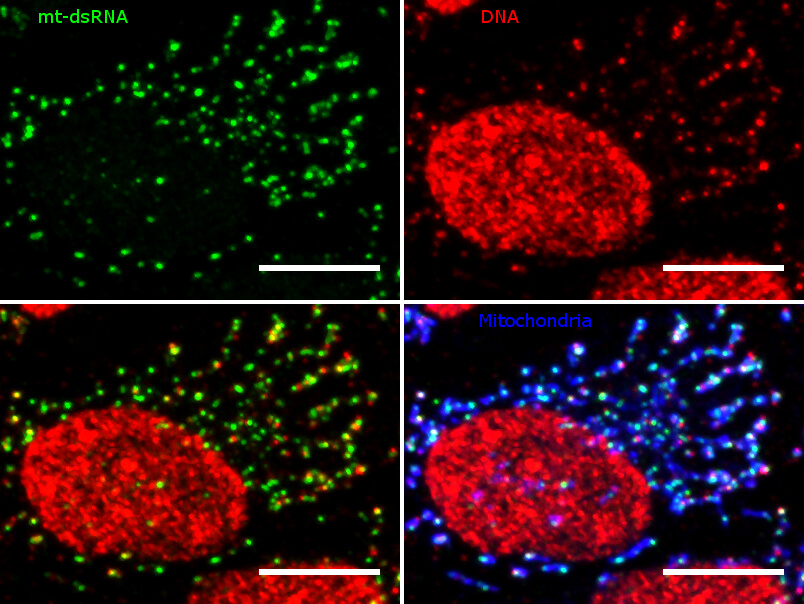
Druga praca dotycząca metabolizmu RNA w ludzkich mitochondriach została opublikowana w czasopiśmie Nature (https://www.nature.com/articles/s41586-018-0363-0). W badaniach tych uczestniczyli: dr Łukasz Borowski (równorzędny drugi autor), prof. Andrzej Dziembowski oraz dr Roman Szczęsny (jeden z autorów korespondujących). W pracy tej zaproponowano, że mitochondria są głównym źródłem dwuniciowego RNA (mt-dsRNA) w komórkach człowieka. Ponadto, stwierdzono, że kompleks białek SUV3-PNPaza, który wcześniej odkryto w IGiB UW, pełni kluczową rolę w regulacji poziomu dwuniciowego RNA mitochondrialnego. Co istotne, wykazano, że w przypadku inaktywacji PNPazy nagromadzony w mitochondriach dwuniciowy RNA może wydostawać się do cytoplazmy, gdzie jest błędnie rozpoznawany jako wirusowy, co w konsekwencji prowadzi do uruchomienia wrodzonej odpowiedzi immunologicznej. Badania te wskazują, że podłoże niektórych chorób człowieka (interferonopatii typu I) może być związane z nieprawidłowym metabolizmem RNA w mitochondriach (komentarz PAP: http://naukawpolsce.pap.pl/aktualnosci/news…).
